N6-甲基腺苷(m6A)是RNA甲基化修饰中最常见的形式,是生信研究的热点方向,相关研究也比较容易发到影响因子在3~10分之间的期刊。m6A调控在肿瘤发生中起着重要作用。经常关注挑圈联靠的小伙伴想必对m6A相关文献套路已经不陌生了。今天我们再分享一个套路:与m6A相关的非编码RNA对肿瘤预后的影响与signature的筛选。
m6A和ncRNA两个发文热点加在一起是否会有更热烈的化学反应呢?冲更高分呢?
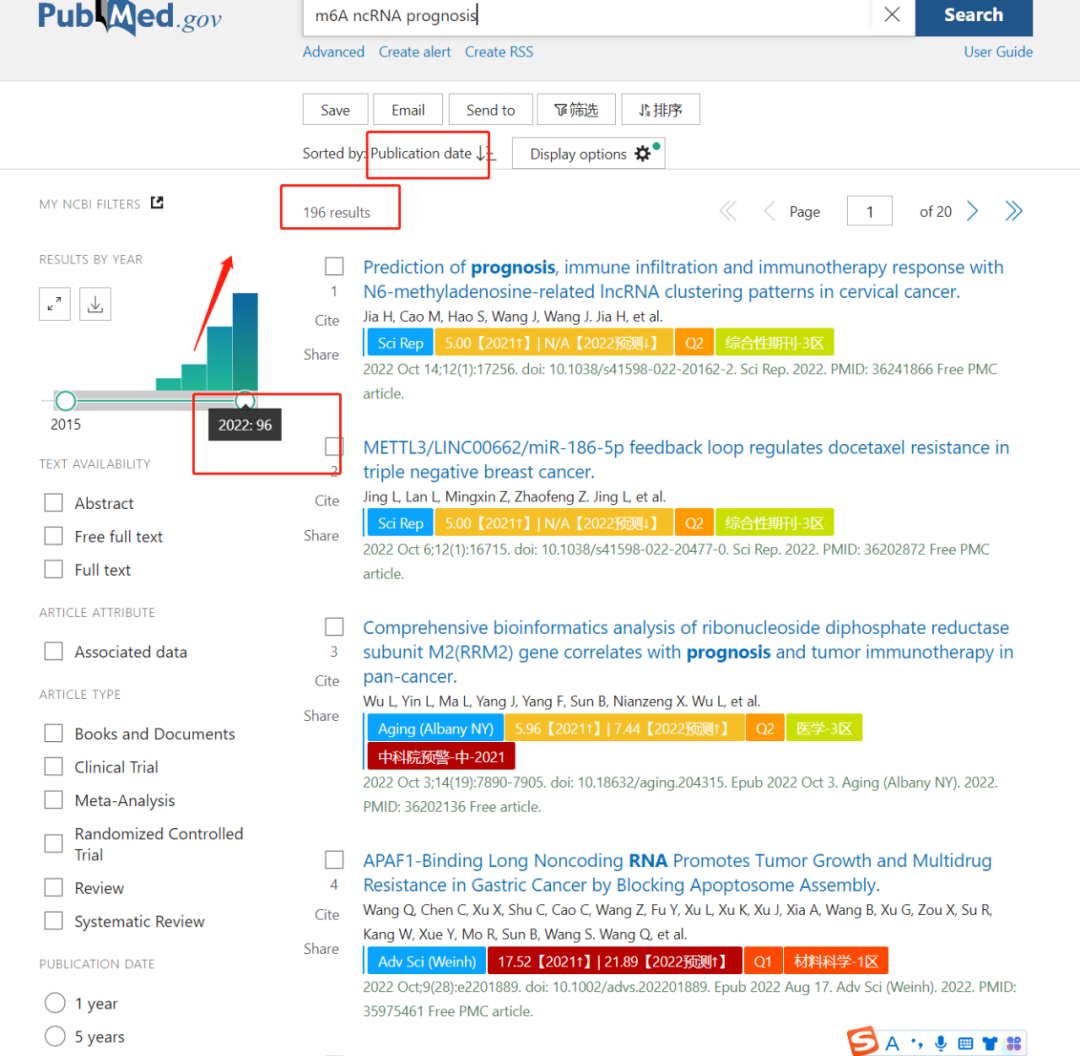
老样子,打开Pubmed,输入m6A ncRNA prognosis,做做市场调查!一共196篇文章,其中接近一半96篇都是还没结束的今年发表的!又是一个新兴热点!按相关性排序,扑面而来一堆Molecular Cancer的40+,天花板可以说是很高!接下来看点画风“正常”的,按出版时间来,很多小伙伴今年新发表的文章还是不错的,保底也有5+,好好做,10+期刊也是不难的。
低至3+5+兜底,高到10+高分文,根据自己需要。
双热点加持!ncRNA种类丰富,有很大挖掘空间!
也只需公共数据库挖掘就能搞定!
光看影响因子是不够的!下面我们就根据一篇10+涉及lncRNA的文献按“挑圈联靠”的套路举例说明一下文章(参考文献里有加粗)。
一、CRC中m6A RNA甲基化调节因子的表达情况(挑)
研究者首先比较了TCGA数据集中19个m6A相关的RNA甲基化调节因子在CRC中的表达。结果发现,除YTHDC2外,所有19个基因在CRC组织中的表达都与正常组织有差异(图1A)。小提琴图显示,CRC组织中的METTL3, WTAP, KIAA1429, RBM15, ZC3H13, YTHDC1, YTHDF1, YTHDF2, HNRNPA2B1, HNRNPC, IGF2BP1, IGF2BP2, IGF2BP3, ALKBH3和FTO与正常组织相比,显著上调;然而CRC组织中的METTL14、YTHDF3和ALKBH5显著下调(图1B)。研究者进一步评估了CNV模式与m6A RNA甲基化调节因子的表达之间的关系。结果表明,m6A调节剂的表达水平与不同的CNV模式有明显的关系。研究结果显示,除了IGF2BP1和IGF2BP3,CNV的增加与较高的基因表达有明显的相关性,而CNV的缺失则导致基因表达的下降(图S1)。研究者确定了19个m6A RNA甲基化调节因子之间的Spearman相关性分析。m6A RNA甲基化调节因子之间的相关性大多为正,KIAA1429和YTHDF3是最正相关的(Cor = 0.81)(图1C)。研究者展示了m6A RNA甲基化调节因子表达与CRC不同临床病理特征之间的关系,包括性别、年龄、肿瘤分期、T期、M期、N期和生命状态(图1D)。二、共识聚类确定了两个ESCC患者的群组(cluster)以及它们与临床病理参数的关系(圈、靠)
使用 "ConsensusClusterPlus "软件包构建了一个由20个m6A RNA甲基化调节因子组成的一致集群。图3A,B显示了共识集群的累积分布函数(CDF)从k=2到9的相对变化,以及CDF曲线下的面积从k=2到9的相对变化,事实证明k=2时,来自TCGA的ESCC队列被分为两个不同的聚类是最合适选择(图3C)。图3D展示了k=2到k=10的跟踪图(tracking plot)。聚类亚群与患者的临床病理参数之间的关系显示,聚类1和聚类2与患者年龄、性别、T期、N期、M期或TNM期之间没有意义(图3E)。
三、WGCNA分析,找到共表达的m6A相关的mrna和lncrna(挑)使用R软件包edgeR,研究者在TCGA数据集中确定了700个lncRNAs和3637个mRNAs进行进一步分析(图S4)。通过WGCNA,将不同RNA归入不同的模块,确定好模块的范围后选择模块关键基因(ME),作为一个给定RNA模块的主要成分。将ME作为一个模块的特定基因表达模式;依此总结基因表达谱,并计算ME与m6A调节因子表达水平的相关性,以确定与m6A相关的lncRNA和mRNA模块。最终,研究者确认了六个lncRNA共表达模块,并评估了它们与12个OS相关的m6A RNA甲基化调节因子的关联(图4A和4B)。选定的不同模块中ME的散点图显示,蓝色、棕色和绿松石色的模块与m6A调节因子有明显的相关性,包括YTHDF1、ALKBH3、ALKBH5和FTO(图4C-4H)。共有544个lncRNAs参与了这三个模块;我们将那些与m6A调节因子相关的lncRNAs定义为m6A相关的lncRNAs。至于mRNAs,有10个模块被确定为进一步分析的对象(图5A和5B)。此外,与其他模块相比,红色和绿松石色的模块与m6A RNA调节因子明显相关(图5C-5H)。这两个模块中共有1292个mRNAs被认为是与m6A相关的mRNAs。五、lncRNA的预后分析(预后m6A相关lncRNA signature的构建和验证)(靠)
利用预后信息,应用单变量Cox回归,从训练集中的544个m6A相关lncRNAs中识别出预后的m6A相关lncRNAs。其中37个m6A相关的lncRNAs与训练集中的CRC个体的OS显著相关。将这37个预后良好的m6A相关lncRNAs的基础上构建了LASSO Cox分析,并产生了m6A相关lncRNAsignature,其中包含24个m6A相关lncRNAs(图S5A和S5B)。根据每个lncRNA的系数(coefficient),计算出每个受影响个体的风险分数。根据风险评分的中位数,将患有CRC的受试者分为低风险组和高风险组。Kaplan-Meier(K-M)生存曲线显示,低风险组的CRC受试者比高风险组的受试者有更好的临床结果:训练组(p = 9.5 × 10-7)(图6A),测试组(p = 2.44 × 10-4)(图S6A),以及TCGA队列总数(p = 1.79 × 10-9)(图S6E)。ROC曲线显示,在训练集(图6B)、测试集(图S6B)和TCGA总队列(图S6F)中,m6A相关的lncRNA signature表现出预测OS的强大准确性。六、mRNA的预后分析(预后m6A相关mRNA signature的构建和验证)(靠)在训练集中,使用单变量Cox回归,75个mRNAs被确定为预后的m6A相关mRNAs。此外,LASSO Cox分析被用来构建一个m6A相关的mRNA signature,涉及33个预后的m6A相关的mRNAs(图S5C和S5D)。根据风险评分的中位数,CRC受试者被分为低风险和高风险组。Kaplan-Meier生存曲线表明,风险分数较高的CRC受试者的临床结果较差(OS时间较短,OS率较低)(图6E)。ROC分析显示,在训练数据集中,m6A相关的mRNA签名具有预测OS的能力(1年曲线下面积[AUC]=0.853,3年AUC=0.891,5年OS=0.892)(图6F)。在测试集(图S6I-S6L)和TCGA总队列(图S6M-S6P)中验证了m6A相关mRNA签名的预后能力。这些结果显示了该特征的强大的OS预测能力。七、m6A相关的lncRNA和mRNA signature是CRC患者的独立预后因素(靠)对TCGA数据的临床病理参数,进行单变量和多变量的Cox回归分析,以确定与m6A相关的lncRNA和mRNA signature的计算的风险分数是否准确预测了CRC个体的预后。结果显示,m6A相关的lncRNA和mRNA signature与OS有明显的相关性,在训练集中,这些特征是OS的独立预后因素(图7A和7B)。这些结论在测试集(图S7A和S7B)和整个TCGA队列(图S7C和S7D)中得到验证。研究者探索风险分数是否与临床病理特征的相关性分析。结果发现,与低风险组相比,高风险组与较高的死亡率和较高的肿瘤分期显著相关,但与年龄和性别没有关系(图7C和7D)。研究者还分析了m6A相关lncRNA和mRNA signature与干细胞表型和药物敏感性之间的关系。研究发现m6A相关的lncRNA和mRNA signature与CRC干细胞表型明显相关(图S8),而且这两个签名中的高危对象可能对药物治疗更敏感(图S9)。多变量Cox分析和风险分数是否与临床病理特征的相关性分析,m6A相关lncRNA和mRNA signature,是否具有独立的预后意义
八、基于m6A相关lncRNA和mRNA signature的列线图构建和校准曲线验证(靠)为了创建一个适用的临床评估工具,以提高预测CRC患者OS的准确性,构建列线图,以预测训练数据集中1年、3年和5年的OS概率(图8A)。图B的校准曲线图显示列线图预测和实际3年OS的比较,列线图的性能在预测3年OS时最好。如图8C所示,在决策曲线分析(DCA)中,列线图在预测相应的1年、3年和5年的OS时,表现出更广泛的阈值概率的净效益。正如DCA所验证的,图8D描述了与m6A相关的lncRNA和基于mRNA signature的风险评分相比,列线图的净临床效益更大。图8E中,KM生存曲线显示,基于列线图的风险评分的中位数,分为两组的CRC受试者的OS。图8F中,与时间相关的ROC曲线显示了在列线图基础上预测1年、3年和5年的OS方面有很好的预后准确性(AUC=0.844、0.855和0.824,分别)(图8F)。根据列线图的风险评分,受试者按中位数分为两组。结果表明,高风险组的受试者与低风险组的受试者相比,其结果明显更差(P < 0.001)(图8E)。使用测试集和TCGA队列总数进行验证,我们验证了同样的结果,如图图S10所示。
一些文献里随处可见的常规分析和更多高级分析也是有的!RNA修饰套路
扫码立即咨询

如果还没有看挑圈联靠之前的m6A等RNA甲基化修饰文章的小伙伴,欢迎进一步阅读:
1+1>2! 这篇7+m6A单细胞代表作模板,直接整高级!
1、Li J, Rao B, Yang J, Liu L, Huang M, Liu X, Cui G, Li C, Han Q, Yang H, Cui X, Sun R. Dysregulated m6A-Related Regulators Are Associated With Tumor Metastasis and Poor Prognosis in Osteosarcoma. Front Oncol. 2020 Jun 2;10:769.(IF=5.738)2、Guo W, Tan F, Huai Q, Wang Z, Shao F, Zhang G, Yang Z, Li R, Xue Q, Gao S, He J. Comprehensive Analysis of PD-L1 Expression, Immune Infiltrates, and m6A RNA Methylation Regulators in Esophageal Squamous Cell Carcinoma. Front Immunol. 2021 May 12;12:669750.(IF=8.786)3、Li W, Gao Y, Jin X, Wang H, Lan T, Wei M, Yan W, Wang G, Li Z, Zhao Z, Jiang X. Comprehensive analysis of N6-methylandenosine regulators and m6A-related RNAs as prognosis factors in colorectal cancer. Mol Ther Nucleic Acids. 2021 Dec 14;27:598-610. (IF=10.183)
—END—
撰文丨三叶虫
排版丨三叶虫
编辑丨三叶虫