糖尿病肾病作为一种具有预防性可早期干预的糖尿病并发症,成为了降低糖尿病死亡率、提升糖尿病患者生存质量的关注重点。为了实现糖尿病肾病的早期诊断,来自香港中文大学的研究团队使用Illumina因美纳甲基化芯片,对糖尿病患者进行了全基因组甲基化检测,以确定与糖尿病肾病相关的表观遗传标记。
鉴于2型糖尿病的发病率在全球范围内显著增长,且发病年龄正在趋于年轻化,这导致了糖尿病终末期肾病(End-Stage Kidney Disease, ESKD)的负担不断增加。而糖尿病肾病(Diabetic Kidney Disease, DKD)具有可预防性,识别有DKD和ESKD进展风险的个体,以便进行早期强化干预已迫在眉睫。尽管已有几种治疗方法可以延缓DKD的进展,可是早期识别与具体治疗方案仍有诸多局限。如何对糖尿病患者中的肾功能障碍高风险人群进行分层成为研究焦点。
目前为了识别可以指导DKD分层的生物标志物,已有研究人员开始尝试使用遗传和其他类型的生物标志物。虽然全基因组关联研究(GWAS)在识别2型糖尿病和其他复杂疾病的遗传标记方面取得了相当大的成功,但在识别与DKD相关基因座方面的进展却不太顺利。根据目前已有的研究发现,常见表观遗传标记中的甲基化标记物是有前途的糖尿病并发症生物标记物。
今年,来自香港中文大学的叶凯文和马志伟在Nature Communications(IF:16.6)发表题为《DNA methylation markers for kidney function and progression of diabetic kidney disease》的论文。在该项研究中,科研人员选择外周血中测量的CpG位点甲基化,已验证其是否可能与肾功能相关,并且构建模型检测该方面信息是否可用于预测2型糖尿病肾功能恶化以达到预后目的。
取样及测序
1271名受试者来自香港中文大学的教学医院威尔斯亲王医院设立的香港糖尿病登记(Hong Kong Diabetes Register, HKDR)项目。根据修改后的欧洲 DiabCare 流程,对糖尿病受试者进行评估。来自HKDR的所有受试者在8小时过夜禁食后均接受了临床评估和实验室检查。
该项目使用了覆盖基因组中约485,000 个 CpG 位点的 Illumina Infinium Human Methylation 450K BeadChip 对血液样本检测,以测量每个样品中的 DNA 甲基化情况。血液样本是在基线评估访视时,让受试者们在禁食状态下采集的。
本研究获得香港中文大学-新界东联网联合临床研究伦理委员会的伦理批准。在入组时获得了所有受试者的书面知情同意书,以收集用于存档和研究目的的临床信息和生物样本。
各个 CpG 位点的甲基化水平与基线肾功能和肾功能下降相关
图1:CpG 甲基化与肾功能之间的关系
a-c:对于基线 eGFR,40 个 CpG 位点呈现表观基因显著,P值<0.05(Bonferroni校正);386个CpG位点则在FDR = 0.05时,具有统计学意义。即在统计学上,基线估算肾小球滤过率与40个CpG位点的甲基化相关,当错误发现率FDR,即在所有结果显著的检验中假阳性所占的比率,为5%时,基线估算肾小球滤过率与386个CpG位点的甲基化相关。
d-f:对于eGFR的斜率,8 个CpG位点呈现表观基因显著,P值<0.05(Bonferroni校正);74个CpG位点则在FDR = 0.05时,具有统计学意义。即在统计学上,随后肾功能下降与8个CpG位点的甲基化相关,当错误发现率FDR,即在所有结果显著的检验中假阳性所占的比率,为5%时,随后肾功能下降与74个CpG位点的甲基化相关。
表1:单位点分析中,CpG 位点的甲基化水平与基线 eGFR 或 eGFR 斜率显著相关在表一中对所有CpG位点均进行单位点分析,p值皆为双侧t检验结果。TSS1500:转录起始位点(TSS)上游 200bp 到 1500bp 之间的区域。在模型系数中,正号表示甲基化水平与基线 eGFR 呈正相关或甲基化水平与eGFR 下降呈负相关,而负号表示相反情况。
在统计学上与基线估算肾小球滤过率关联性最强的 CpG 位点是位于 19 号染色体 ZNF788和ZNF20之间的cg17944885。在统计学上与随后肾功能下降关联性最强的 CpG 位点是位于 21 号染色体的一个CpG 岛上的cg10272901。此外,CpG 位点cg04983687、cg01676795、cg22460173、cg15826891和cg02950701在其他学者的研究中也有过相关报道。
多位点模型捕捉多个人群中 DNA 甲基化和肾功能之间的关系表2:基线 eGFR 最终多位点模型中的 CpG 位点
模型中系数为零的位点是被选择作为拉索回归的输入位点。TSS200:转录起始位点(TSS)与其上游 200bp 之间的区域。Single-site corrected P value: 全表观基因组关联研究结果中的Bonferroni 校正 P 值。
表3:eGFR 斜率最终多位点模型中的 CpG 位点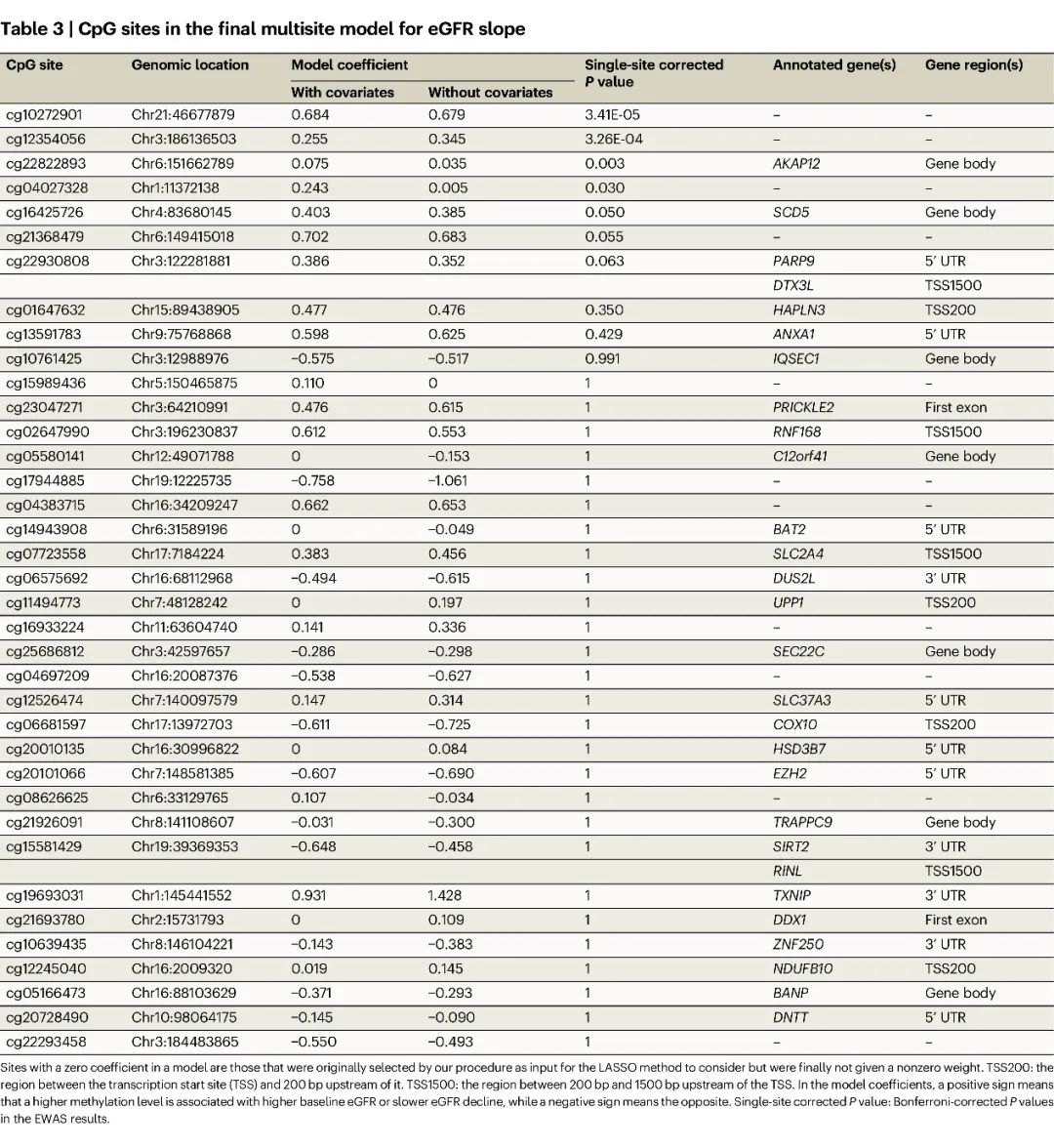
由于CpG 位点的基因组空间依赖性等导致CpG位点间可能彼此强烈相关,故单位点模型存在天然缺陷。为解决这一缺陷,本研究开发了一种多位点方法:同时考虑所有 CpG 位点,并选择其中的一个子集来创建最佳模型来推断基线 eGFR 或 eGFR 斜率。表2、表3:在这些“最终”的多位点模型中,分别包含 64 个和 37 个 CpG 位点,用于预测基线 eGFR 和 eGFR 斜率。表4:根据主要队列的数据构建并应用于主要队列或美洲原住民队列的多位点模型的性能(使用两个队列均可用的 CpG 位点进行训练)PCC:Pearson 相关系数;SCC:Spearman 相关系数;MAE:平均绝对误差。“CpG 位点”列显示了在拉索回归中选择的位点数量,其中一些最终被 拉索回归赋予了零权重。a-b:对于基线 eGFR 和 eGFR 斜率,实际值和最终模型推断的值的 Pearson 相关性分别为 0.806 和 0.635。即在统计学上,基线估算肾小球滤过率和随后肾功能下降的实际值与于CpG 位点呈极强相关与最终模型推断值呈强相关。c-d:有协变量的模型的性能优于没有协变量的模型。e-h:使用包含326 名患有2 型糖尿病的美国原住民受试者的独立队列血液样本进行的全基因组甲基化检测结果表明,本实验的多位点模型面对存在种族差异的样本时,在预测2型糖尿病的基线eGFR和eGFR下降方面也具有良好的性能。。单位点和多位点分析中所选位点的近端基因具有潜在的肾功能
图3:在我们的单位点和多位点分析中支持已确定的 CpG 位点附近基因的功能意义图3: 单位点和多位点分析确定了 348 个(对应于 358 个基因)与基线 eGFR 相关且接近(1 kb 内)注释基因的 CpG 位点,其中的 228 个CpG 位点(对应于 215 个基因,占比65.5%)在已知的过往研究中至少有一项报道,与人类全基因组相比,相当于 1.25 倍富集(超几何检验 P = 3.78 × 10-6) 。对于 eGFR 斜率,76 个 CpG 位点中的 51 个(对应 89 个基因中的 52 个,占比67.1%)在已知的过往研究中至少有一项报道为与肾功能潜在相关,与人类全基因组相比,相当于 1.21 倍富集(超几何检验 P = 0.03,)。多部位模型推断的 eGFR 斜率可以预测未来的肾衰竭
图4:当考虑所有患者时,协变量多部位模型的接收者-操作者特征下的面积(area under the receiver–operator characteristic, AUROC) 为 0.94,AUPR(精确回忆曲线下面积)为 0.73。当排除基线 eGFR<30 ml/min/1.73 m 2的患者(5 年内发生 ESKD 的风险非常高)时,协变量模型的 AUROC 为 0.88,AUPR 为 0.36。在这两种情况下,即使没有临床协变量,本研究模型的性能也与临床方程的性能相当,并且临床协变量的包含进一步增强了模型的性能。
在此研究中科研人员主要发现了:
(2) 先前未识别的 CpG 位点的甲基化水平与基线 eGFR 相关;(3) 一组八个先前未识别的 CpG 位点与 eGFR 下降率相关;(4) 可以使用基线 eGFR 的甲基化数据构建预测模型,并在独立的2型糖尿病队列中复制 eGFR 的下降;(5) 先前未识别的 CpG 位点和预测模型中包含的近端基因与肾脏疾病发病机制相关的通路有关;(6) 构建的预测模型可以实现与结合临床危险因素的模型相当的预测。不过本实验还是存在一定局限性:尽管验证结果表明研究所开发的模型同样适用于不同临床环境的糖尿病患者,但本研究是在 DKD 进展风险相对较高的中国患者中进行的,研究对象终究存在局限性。此外,本研究的甲基化特征是基于循环白细胞的甲基化变化,而不是肾脏内的甲基化变化,这也可能导致实验结果与真实结果有所偏差。但相较于以往的研究,该实验不仅在地域人种上不同——以往的 EWAS 研究主要在欧洲人群中进行,而且样本量相对较大。在研究中确定的与糖尿病肾病关联性最强的 CpG 位点 cg17944885 在过往的 EWAS 研究中均有报告与肾功能相关。意味着在健康人群中与肾功能相关的甲基化改变也可能适用于2型糖尿病患者。与肾功能下降相关度强的甲基化标志物与基线估算肾小球滤过率相关度强的甲基化标志物重叠度小,可考虑作为判断糖尿病肾病预后的生物标记。总的来说,实验中发现的强关联 CPG 位点大多位于基因体中。而且与糖尿病人肾功能下降相关的74个 CpG 位点在以往对健康人群进行的肾功能研究中未被报道过,意味着针对糖尿病患者进行糖尿病肾病进行精准医疗存在可能。此外,在此项研究中主要采用了 Illumina InfiniumTM Human Methylation 450K Beadchip 对样本进行了甲基化检测。去年12月,因美纳发布了甲基化芯片产品的升级版本 InfiniumTM Methylation EPIC v2.0 BeadChip。全新上市的 InfiniumTM Methylation EPIC v2.0 BeadChip 包括超过 935K CpG ,覆盖增强子、CTCF结合位点和常见癌症驱动突变,并为甲基化分类方法用户优化了拷贝数变异(CNV)检出。InfiniumTM Methylation EPIC v2.0 BeadChip 可以低廉的单样本成本产出高度精准的DNA甲基化数据,并兼容从FFPE组织样本提取的DNA。相比于 WGBS、RRBS 和靶向甲基化测序,用 EPIC v2.0 获取全基因组DNA甲基化数据的成本显著降低,更适合大型队列研究中对大规模样本行进表观遗传标志物筛查。本论文的原文链接:https://www.nature.com/articles/s41467-023-37837-7
