3)研究对象:直肠癌(rectal cancer)
另外,本文的代码已开源,详见:https://github.com/KatherLab/dl-mri.
预后模型(I/C):Vision Transformer模型构建的影像组学signature结局指标(O):病人的无疾病生存期(DFS)和总生存期(OS)研究目的:基于更为强大的深度学习特征提取器Vision Transformer(ViT),提取T2-weighted MRI影像特征构建影像标志物,用于直肠癌病人的风险指标。
协变量(Z):年龄、性别、肿瘤位置、T分期、N分期、癌胚抗原(CEA)水平、是否接受放疗临床问题:如何开发有效的方法预测直肠癌病人的预后。
目前方法不足:目前临床上主要通过TNM分期和肿瘤退缩分级(Tumor regression grading, TRG)来预测预后,也有一些工作借助预定义的影像特征来完成预测任务,但是:1)对于使用TNM分期的方法,属于同一分期的肿瘤之间可能也存在异质性;2)基于TRG的方法需要进行肿瘤组织的采样,是有创的,无法在诊断过程中完成;3)预定义影像特征的计算依赖于肿瘤区域的精细化标注,同时提取肿瘤信息的能力相对有限。本文解决方式:作者基于T2加权的MRI影像,选取特征提取能力更为强大的Vision Transformer(ViT)提取肿瘤影像特征,进而构建直肠癌的预后影像标志物,以期为临床诊治决策提供参考。
我们先来看一下流程图 (Figure 2A),研究的主要步骤为:② 将来自中国医院的病人划分为训练集、验证集、内部测试集,来自德国医院的病人作为外部测试集③ 关注T2WI的MRI影像,根据肿瘤标注选取面积最大的层面④ 将肿瘤影像输入到Vision Transformer中,获得deep learning score第一部分:文章方法部分解读
纳入标准
① 经组织病理学证实的直肠腺癌
② 有新辅助治疗开始前1周内的治疗前MRI影像
③ MRI评估显示下边缘在肛门边缘上方15cm内的肿瘤
④ 术前临床分期在T3或更高,或者是非N0分期,或者是MRI评估显示环周切缘受侵
⑤ 在新辅助治疗后才进行全直肠系膜切除术
注:环周切缘受侵(circumferential resection margin involvement)相关内容可以参考:https://www.sohu.com/a/728641914_121124542
与训练集相同,区别在于这部分病人没有接受新辅助治疗
与训练集的①-③相同
① 病人有并发恶性肿瘤或既往抗癌治疗史
② 未完成新辅助治疗或未进行根治性手术
③ MRI数据缺失或图像质量较差
④ 临床随访数据缺失或不完整
详见Figure 1,图中A、B、C分别对应训练+验证集、内部测试集和外部测试集:治疗方案:不限制(训练&验证集、外部测试集的病人接受了新辅助治疗,而内部测试集的病人未接受新辅助治疗);层厚:来自中国医院的层厚为5mm,来自德国医院的层厚为1.5-6mm;注:这里构建深度学习模型分别预测DFS和OS,以二者各自的损失函数按1:1的比例求和,作为总的损失函数。最终,共纳入3个队列的895名患者,并从中获得T2WI影像数据、临床特征及随访数据:•训练集&验证集:来自中国医院,队列按7:3划分为训练和验证集,训练集507例患者,验证集218例患者,用于模型训练和最佳参数选取•内部测试集:来自中国医院,112例患者,用于模型在同中心的内部验证•外部测试集:来自德国医院,58例患者,104个病灶,用于模型在不同中心的外部验证
对于来自中国中心的影像,分割过程由两位具有 15年影像解读经验的放射科医师执行,二者的操作是独立的。对于来自德国中心的影像,分割过程由两位分别具有5年和3年经验的外科医生执行,并由一位具有10年结直肠癌阅片经验的高级放射科医师验证。所有的图像分割均使用ITK-SNAP软件完成,每一个标注都由一位具有至少8年直肠癌图像处理经验的专家进行审核。模型输入的图像为T2WI影像经分割后肿瘤面积最大的层面。本研究使用深度学习策略提取影像特征。与常见的深度学习+影像组学的工作不同,本研究使用了Vision Transformer(ViT)模型来提取影像特征。模型会在第6节进行介绍。模型的输入是肿瘤区域的影像,区域边界为肿瘤区域向外扩展10个像素。肿瘤区域的影像会缩放到224x224的大小输入模型中。由于深度学习模型是端到端(end-to-end)的形式,特征提取会在训练中自动加以优化,而非经由固定的公式计算得出。ViT模型在经过训练后,其模型参数具有特征选择作用。这一过程也经由端到端的学习实现,没有显式的特征选择步骤。本研究使用的ViT模型具体是ViT-B/16架构,其中B表示Transformer的编码器尺寸,对应于base(其他尺寸还有H-huge和L-large)。ViT模型的输入是patch的形式,即将原始图像以网格的形式切分成若干子图,然后每个子图分别编码为token的形式输入transformer模型中,这里的16指的是将图像切片成若干个patch后每个patch的尺寸。这里涉及到了一些ViT相关的知识,感兴趣的读者推荐查看下方链接做更多的了解,不感兴趣可以将这里当做一个黑盒模型,不会影响对文章架构的理解,这里的核心在于从影像中提取特征然后输出deep-learning-score,和CNN是一样的。https://blog.csdn.net/weixin_42392454/article/details/122667271 本研究使用的ViT模型使用在ImageNet-21K数据集预训练的参数进行初始化,模型训练的一些细节:- 防过拟合策略:Early stop(验证集loss连续10个epoch不下降则停止训练)
这里损失函数采用的是部分负对数似然函数(Negative partial log-likelihood),计算公式如下:这里作者对公式的解读较为直接,我们在此处略微进行深入挖掘,以帮助读者理解损失函数的设计思想。式中, 函数表示深度学习模型,θ是模型参数,Xi 是输入样本,那么
函数表示深度学习模型,θ是模型参数,Xi 是输入样本,那么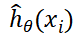 就是样本i输入模型获得的输出。Ei=1表示所有确定死亡的样本集合(而不是删失数据),
就是样本i输入模型获得的输出。Ei=1表示所有确定死亡的样本集合(而不是删失数据), 表示所有生存时间大于样本i的样本集合。这个公式乍一看非常难懂,但实际上,它来源于经典的Cox模型。Cox比例风险模型的公式如下:
表示所有生存时间大于样本i的样本集合。这个公式乍一看非常难懂,但实际上,它来源于经典的Cox模型。Cox比例风险模型的公式如下: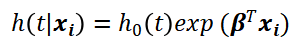
式中的βT即为变量的系数向量。对于一个样本i,在时刻t发生事件的概率为样本i在时刻t的风险与所有事件晚于i的样本风险之和的比值,而与事件发生早于i的样本无关,即:
部分对数似然函数即为所有发生事件样本概率的乘积再取对数: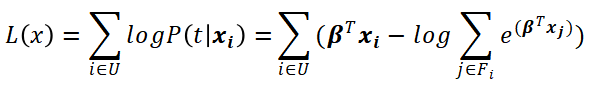
模型优化的目标应当是使得似然函数最大化,因此可以采用部分对数似然函数的负值作为损失函数。对于深度学习模型,将参数β替换为模型映射函数 即可,这样就得到了上文中给出的损失函数形式。需要注意的是,由于
即可,这样就得到了上文中给出的损失函数形式。需要注意的是,由于 的存在,计算损失函数是既要输入生存时间,也要输入样本的存活状态。最后,为了同时考虑两种类型的生存数据,最终的损失函数对DFS和OS采用1:1的比例进行加权求和。模型训练完成后,对于每个样本的输出即为该样本的DL-model-computed score,由于预测的目标为病人生存风险,这里也称为Risk Score。原文的Figure2B给出了使用ViT模型预测风险的全流程:本研究对ViT模型及其输出的DL-model-computed score的预测和预后性能进行了全面的评估。除了这一分数,本研究还结合新辅助治疗前的癌胚抗原(carcinoembryonic antigen,CEA)水平,使用XGBoost模型构建了一个多模态风险分数(multi-modal risk score),并对这个分数的预测和分层能力进行了一系列的评估。XGBoost模型使用训练集和验证集完成模型训练,并在内部和外部测试集上验证模型效果,如果病人的CEA数据缺失,则置零。原文的Figure2C给出了本研究对两个风险分数评估的流程示意图。
的存在,计算损失函数是既要输入生存时间,也要输入样本的存活状态。最后,为了同时考虑两种类型的生存数据,最终的损失函数对DFS和OS采用1:1的比例进行加权求和。模型训练完成后,对于每个样本的输出即为该样本的DL-model-computed score,由于预测的目标为病人生存风险,这里也称为Risk Score。原文的Figure2B给出了使用ViT模型预测风险的全流程:本研究对ViT模型及其输出的DL-model-computed score的预测和预后性能进行了全面的评估。除了这一分数,本研究还结合新辅助治疗前的癌胚抗原(carcinoembryonic antigen,CEA)水平,使用XGBoost模型构建了一个多模态风险分数(multi-modal risk score),并对这个分数的预测和分层能力进行了一系列的评估。XGBoost模型使用训练集和验证集完成模型训练,并在内部和外部测试集上验证模型效果,如果病人的CEA数据缺失,则置零。原文的Figure2C给出了本研究对两个风险分数评估的流程示意图。
Figure S1展示了本文使用的ViT-B/16模型和其他深度学习模型的参数量差别ViT模型的参数量相比常见的深度学习模型(基于卷积神经网络)有了明显提升,这可能是模型性能提高的基础。这里顺便说一下,最近非常流行的ChatGPT等大模型的基础就是Transformer。Table S2比较了ViT-B/16模型和各个baseline模型预测生存期的C-index在验证集上,ViT模型预测的C-index要显著高于其他baseline模型的预测结果,说明模型对于生存风险的预测能力强,且泛化性良好。Figure S2、S3比较了ViT模型预测生存状态(二分类)的AUCViT模型预测1年、3年和5年生存状态的能力较强,除了在外部测试集上预测3年和5年DFS的结果(Figure S3右下角子图),其他结果说明了模型的泛化性能也较好。作者在训练集上选取DL-model-computed score (为方便表示,后续统一简称为DLS)的阈值(定为-1.037),之后在训练集和其他三个数据集中分别以该阈值对病人分组,使用K-M图以及log-rank检验的p值判断组间是否存在显著差异。此外,在根据病人临床特征(比如性别、年龄、分期等)划分的病人亚组中也重复了该实验。Figure 3A展示了DLS针对训练集和验证集病人OS和DFS的分层情况在训练集和验证集中,DLS可以显著区分生存风险明显有差异的病人。Figure 4展示了DLS针对内部和外部测试集病人OS和DFS的分层情况在内部和外部测试集中,DLS可以显著区分总生存(OS)风险明显有差异的病人,对于DFS,DLS在内部测试集中区分能力较好,而在外部测试集中的扩展能力有限。Figure S4、S5分别展示了DLS针对内部测试集病人亚组的DFS和OS分层情况对于绝大多数临床特征划分的病人亚组,使用DLS分层获得的病人分组间均存在显著的生存差异。Figure S6展示了DLS针对验证集病人处于TRG0/1级亚组的DFS和OS分层情况即便对于预后相对良好的TRG0/TRG1亚组,使用DLS分层获得的病人分组间也存在显著的OS差异。(TRG: tumor regression grade)。Figure S7展示了DLS与病人从放疗治疗中获益之间的关联对于OS,放疗治疗(RT)不会提升病人的总生存期;对于DFS,放疗治疗(RT)能够降低病人的复发风险,同时对于DLS判定的低风险组,放疗治疗能够提高病人的无病生存率,意味着对于该组病人采用放疗治疗有助于降低直肠癌的复发风险。Figure 3B展示了内部测试集中DLS的分布情况与典型案例图左侧是DLS的分布情况,右侧分别为对应的影像、grad-cam可视化、attention map可视化。上节中介绍了DLS的阈值为-1.307,这里分布图中据此标注了“better prognosis”和“worse prognosis”。对于grad-cam和attention map可视化,作者并未做更多的说明,这里可能以展示为主。读者如果对深度学习可视化有兴趣的话,grad-cam可以参考文献1,attention map可以参考:https://www.zhihu.com/question/401506148/answer/3209394886
作者将DLS(二值化为高/低)和其他临床变量一同进行多变量回归,包括年龄、性别、肿瘤位置、T分期、N分期和新辅助治疗前后的癌胚抗原(carcinoembryonic antigen,CEA)水平。Figure 5、S8展示了多变量回归的HR和p-value在训练集、验证集和内部测试集合上,高DLS(图中的Risk)通常是生存期的显著不良预后因素,结果的泛化性较好。
Figure 6A、Table S5展示了CEA、DLS及其组合成的多模态风险分数预测生存期的C-indexDLS预测生存期的C-index显著好于CEA,二者结合的多模态风险分数在大多数数据集中相较DLS有了进一步的提升。Figure 6B展示了训练集和内部测试集上多模态风险分数对病人预后的分层能力多模态风险分数对于病人OS和DFS的分层能力较好,能够有效区分出高风险和低风险病人。
本文基于治疗前T2WI的直肠癌影像,使用Vision Transformer(ViT)模型提取肿瘤影像特征,开发了直肠癌预后的影像标志物,并在下游任务中验证了其有效性。本研究发表在了影像顶刊Radiology。它成功的原因在哪里呢?我认为原因在以下几点:①螃蟹吃得早。在大部分研究开始使用CNN这类深度学习模型的时候,本文是当下为数不多的使用ViT模型的工作,在模型设计上具有先发优势。这也说明了平时多涉猎一些机器学习的前沿还是有好处的。②数据量大。本研究收集了中国和德国两家医院的上千例病人数据,筛选后有895例病人入组。做大了跨中心、跨人种的分析。③全面性好。本研究基于收集到的病人数据,把能做的实验基本都做了,包括建模、预测、分组、多组学结合等,尤其是亚组分析做的非常细致。④套路应有的步骤,文章基本都具备了,实现了比较完整的论述。
从文章总体的内容来看,做的比较规范、系统,质量佳,是个学习的好模板。然而,本文也有一些可提升之处:①临床意义不够明确。由于ViT模型需要的训练样本较多,本文并没有限定病人接受的治疗方式以及肿瘤所处的分期,临床应用场景不够细致,整体来说做的比较粗糙。②本研究使用的是2D肿瘤数据,这样的好处是可以快速使用ViT预训练参数,但却丢失了肿瘤在垂直方向的信息。③仅用了T2WI的影像,而没有考虑其他模态的影像。④对结果的解释不够。比如图6中将病人分为三组,并没有对每个组病人的特性和实验结果的临床意义做深入挖掘,导致文章中很多表述均为实验结果的叙述,而没有论点。⑤没能进一步与Radiomic signature (RS) 进行比较。本研究的一小部分实验结果并不是非常理想,这种情况下应当考虑和传统影像组学方法所获得的RS进行对比,如果比RS表现好,则更能说明方法的有效性。本研究将Transformer模型引入到影像组学的分析中来,具有方法上的创新性,这也是其能发到Radiology杂志的重要原因之一。影像组学向来和机器学习有着非常紧密的结合,对于关注影像组学的我们也可以考虑平时了解一下机器学习的前沿,这样有助于优化建模的方法。它山之石,可以攻玉。好啦,本期的文献分享到这里,对于追求顶刊的小伙伴是不是又一次的冲击呢?继续跟紧君莲数据库的平鑫而论专栏,我们下期见吧~~[1] Selvaraju, R et al., Grad-CAM: Visual explanations from deep net- works via gradient-based localization, 2017 IEEE International Conference on Computer Vision (ICCV) : 618-626.[2] Jiang, X et al., An MRI Deep Learning Model Predicts Outcome in Rectal Cancer, Radiology 2023, 307(5): e222223.